- Replicação do ADN em Bactérias
- Replicação do ADN em eucariotas
- Replicação do ADN de elementos extracromossómicos: plasmídeos e vírus
Objectivos de aprendizagem
- Explicar o significado de replicação semiconservativa do ADN
- Explicar por que razão a replicação do ADN é bidirecional e inclui uma cadeia principal e uma cadeia secundária
- Explicar porque se formam os fragmentos de Okazaki
- Descrever o processo de replicação do ADN e as funções das enzimas envolvidas
- Identificar as diferenças entre a replicação do ADN em bactérias e eucariotas
- Explicar o processo de replicação de círculos rolantes
A elucidação da estrutura da dupla hélice por James Watson e Francisco Crick em 1953 deu uma pista sobre como ADN é copiado durante o processo de replicação A separação das cadeias da dupla hélice forneceria dois modelos para a síntese de novas cadeias complementares, mas a forma exacta como as novas moléculas de ADN eram construídas ainda não era clara. Num modelo, replicação semiconservativa Durante a replicação do ADN, as duas cadeias da dupla hélice separam-se e cada uma delas serve de molde a partir do qual a nova cadeia complementar Após a replicação, cada ADN de cadeia dupla inclui uma cadeia parental ou "antiga" e uma "nova". Foram também sugeridos dois modelos concorrentes: conservador e dispersivo, que são apresentados na Figura 1.
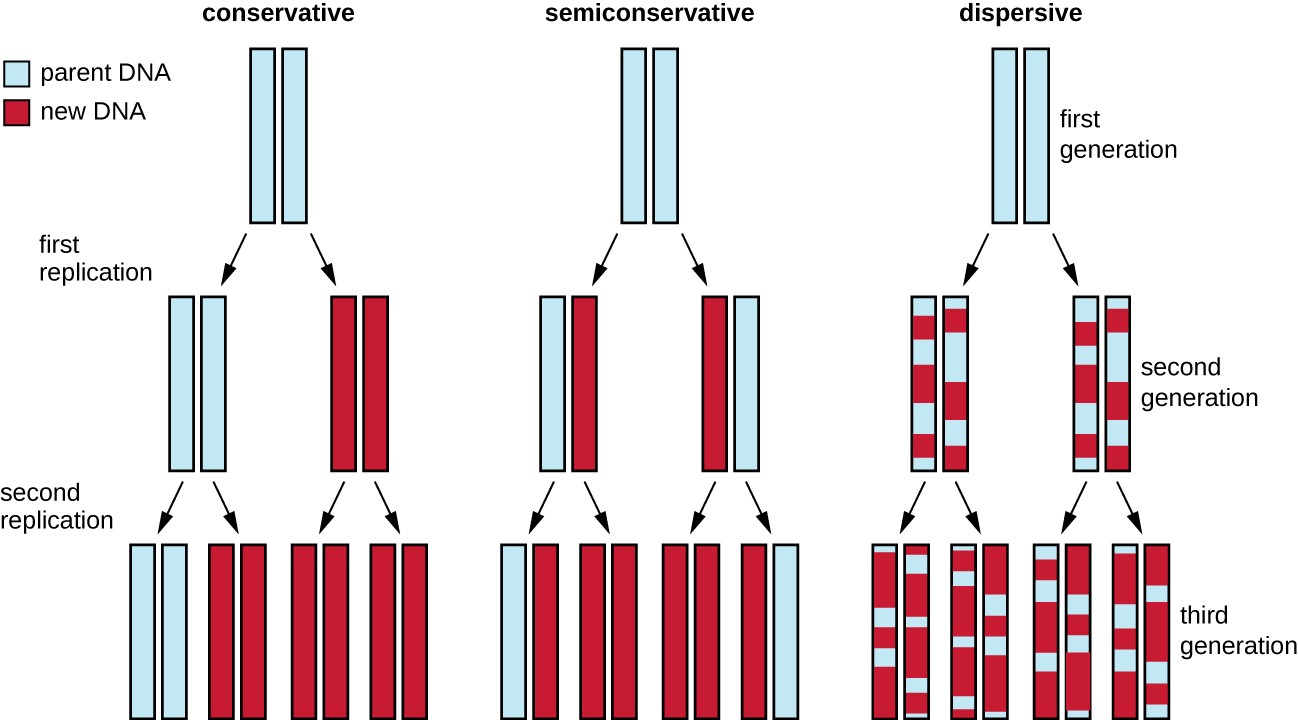
No modelo conservador, as cadeias parentais de ADN (azul) permanecem associadas numa molécula de ADN, enquanto as novas cadeias filhas (vermelho) permanecem associadas em moléculas de ADN recém-formadas. No modelo semiconservador, as cadeias parentais separam-se e dirigem a síntese de uma cadeia filha, sendo cada molécula de ADN resultante um híbridoNo modelo dispersivo, todas as cadeias de ADN resultantes têm regiões de ADN parental de cadeia dupla e regiões de ADN filha de cadeia dupla.
Mateus Meselson (1930-) e Franklin Stahl (1929-) concebeu uma experiência em 1958 para testar qual destes modelos representa corretamente a replicação do ADN (Figura 2). E. coli durante várias gerações num meio que continha um isótopo "pesado" de azoto (15N) que foi incorporado nas bases azotadas e, eventualmente, no ADN. Este isótopo rotulou o ADN parental. E. coli A cultura foi então transferida para um meio contendo 14N e deixada crescer durante uma geração. As células foram colhidas e o ADN foi isolado. O ADN foi separado por ultracentrifugação, durante a qual o ADN formou bandas de acordo com a sua densidade. Seria de esperar que o ADN cultivado em 15N formasse uma banda numa posição de densidade mais elevada do que o cultivado em 14N. Meselson e Stahl observaram que, após umaA única banda observada na geração de crescimento em 14N estava numa posição intermédia entre o ADN das células cultivadas exclusivamente em 15N ou em 14N, o que sugeria um modo de replicação semiconservativo ou dispersivo. Algumas células foram deixadas crescer durante mais uma geração em 14N e centrifugadas novamente. O ADN colhido das células cultivadas durante duas gerações em 14N formou duas bandas: uma banda de ADN estava na posiçãoEstes resultados só poderiam ser explicados se o ADN se replicasse de forma semiconservativa, pelo que os outros dois modelos foram excluídos. Como resultado desta experiência, sabemos agora que, durante a replicação do ADN, cada uma das duas cadeias que constituem a dupla hélice serve de molde a partir do qual são criadas novas cadeiasAs moléculas de ADN resultantes têm a mesma sequência e são divididas em partes iguais nas duas células filhas.
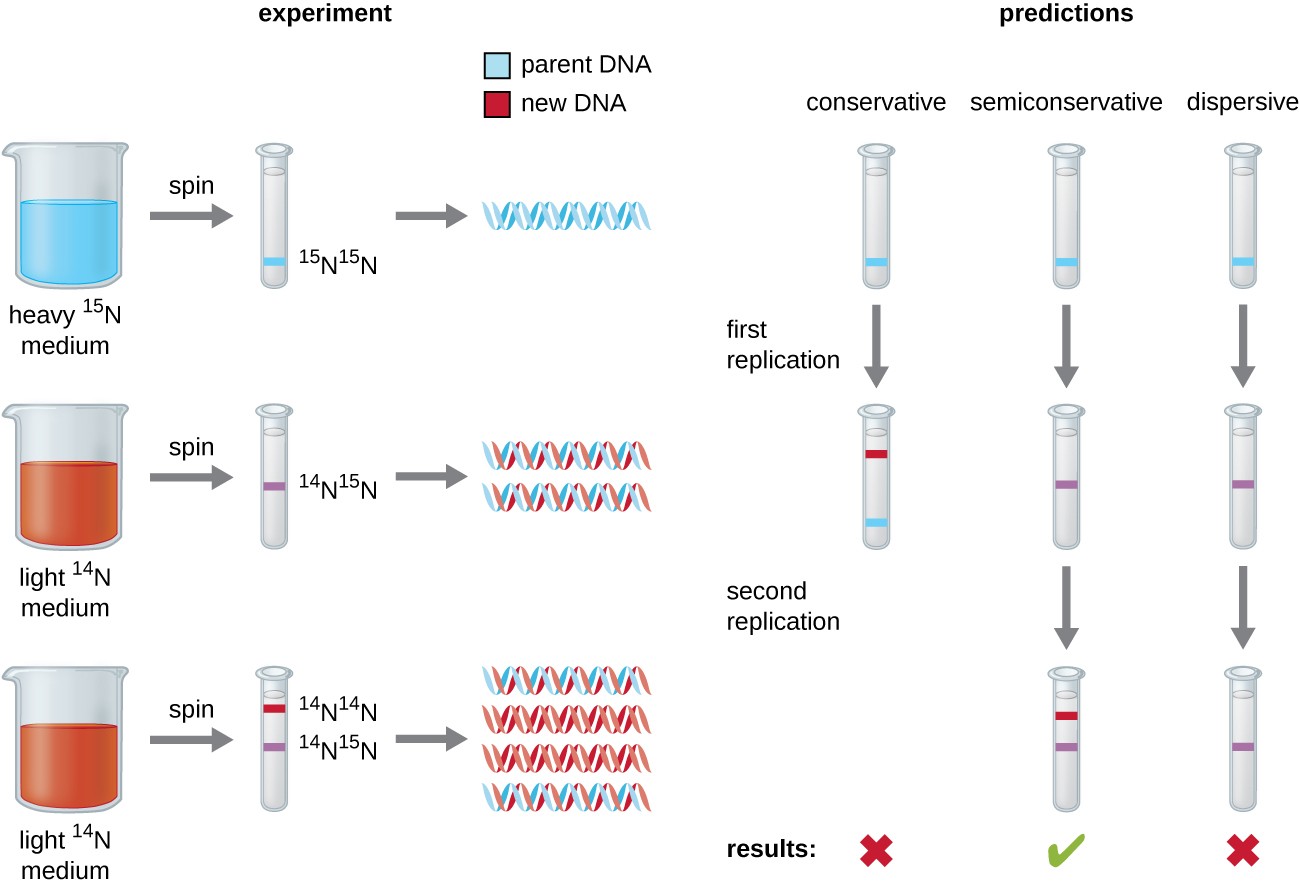
Figura 2. Meselson e Stahl fizeram experiências com E. coli O ADN cultivado em 15N (banda azul) era mais pesado do que o ADN cultivado em 14N (banda vermelha) e sedimentou a um nível inferior na ultracentrifugação. Após uma ronda de replicação, o ADN sedimentou a meio caminho entre os níveis de 15N e 14N (banda roxa), excluindo o modelo conservador de replicação.Estes dados apoiam o modelo de replicação semiconservativo.
Pensar no assunto
- Qual teria sido a conclusão da experiência de Meselson e Stahl se, após a primeira geração, tivessem encontrado duas bandas de ADN?
Replicação do ADN em Bactérias
A replicação do ADN tem sido bem estudada nas bactérias, principalmente devido ao pequeno tamanho do genoma e aos mutantes disponíveis. E. coli tem 4,6 milhões de pares de bases (Mbp) num único cromossoma circular e é replicado na sua totalidade em cerca de 42 minutos, partindo de uma única origem de replicação e prosseguindo à volta do círculo bidireccionalmente (ou seja, em ambas as direcções). Isto significa que são adicionados cerca de 1000 nucleótidos por segundo. O processo é bastante rápido e ocorre com poucos erros.
A replicação do ADN utiliza um grande número de proteínas e enzimas (Quadro 1).
| Tabela 1: A maquinaria molecular envolvida na replicação do ADN bacteriano | |
|---|---|
| Enzima ou Fator | Função |
| DNA pol I | A atividade da exonuclease remove o iniciador de ARN e substitui-o por ADN recém-sintetizado |
| DNA pol III | Principal enzima que adiciona nucleótidos na direção 5′ a 3′ |
| Helicase | Abre a hélice do ADN ao quebrar as ligações de hidrogénio entre as bases azotadas |
| Ligase | Sela as lacunas entre os fragmentos de Okazaki na cadeia de atraso para criar uma cadeia de ADN contínua |
| Primase | Sintetiza os primers de ARN necessários para iniciar a replicação |
| Proteínas de ligação de cadeia simples | Ligam-se ao ADN de cadeia simples para impedir a ligação de hidrogénio entre as cadeias de ADN, reformando o ADN de cadeia dupla |
| Braçadeira deslizante | Ajuda a manter o DNA pol III no lugar quando os nucleótidos estão a ser adicionados |
| Topoisomerase II (DNA girase) | Relaxa o cromossoma em espiral para tornar o ADN mais acessível para o início da replicação; ajuda a aliviar a tensão no ADN quando este se desenrola, provocando quebras e voltando a selar o ADN |
| Topoisomerase IV | Introduz uma quebra de cadeia simples nos cromossomas concatenados para os separar uns dos outros e, em seguida, volta a selar o ADN |
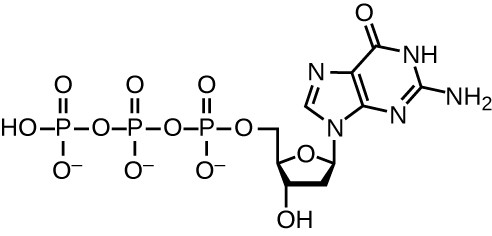
Figura 3: Esta estrutura mostra o desoxirribonucleótido trifosfato de guanosina que é incorporado numa cadeia de ADN em crescimento através da clivagem dos dois grupos fosfato terminais da molécula e da transferência de energia para a ligação fosfato-açúcar. Os outros três nucleótidos formam estruturas análogas.
Um dos principais intervenientes é a enzima ADN polimerase Nas bactérias, são conhecidos três tipos principais de ADN polimerases: ADN pol I, ADN pol II e ADN pol III. Sabe-se agora que a ADN pol III é a enzima necessária para a síntese de ADN; a ADN pol I e a ADN pol II são principalmente necessárias para a reparação. A ADN pol III adiciona desoxirribonucleótidos, cada um deles complementar a um nucleótido da cadeia molde, um a um ao grupo 3′-OH da cadeiaA adição destes nucleótidos requer energia. Esta energia está presente nas ligações de três grupos fosfato ligados a cada nucleótido (um nucleótido trifosfato), à semelhança da energia armazenada nas ligações fosfato do trifosfato de adenosina (ATP) (Figura 3). Quando a ligação entre os fosfatos é quebrada e o difosfato é libertado, a energia libertada permite aformação de uma ligação fosfodiéster covalente por síntese de desidratação entre o nucleótido que chega e o grupo 3′-OH livre na cadeia de ADN em crescimento.
Iniciação
O início da replicação ocorre numa sequência específica de nucleótidos chamada origem da replicação onde várias proteínas se ligam para iniciar o processo de replicação. E. coli tem uma única origem de replicação (como a maioria dos procariotas), chamada oriC A origem da replicação tem aproximadamente 245 pares de bases e é rica em sequências de adenina-timina (AT).
Algumas das proteínas que se ligam à origem da replicação são importantes para tornar as regiões de cadeia simples do ADN acessíveis à replicação. O ADN cromossómico é normalmente enrolado em torno de histonas (em eucariotas e archaea) ou proteínas do tipo histona (em bactérias), e é superenrolado No entanto, enzimas chamadas topoisomerases alteram a forma e o enrolamento do cromossoma. Para que a replicação do ADN bacteriano se inicie, o cromossoma enrolado é relaxado por topoisomerase II , também designado por DNA girase Uma enzima chamada helicase separa as cadeias de ADN, quebrando as ligações de hidrogénio entre os pares de bases azotadas. Recorde-se que as sequências AT têm menos ligações de hidrogénio e, por isso, têm interacções mais fracas do que as sequências guanina-citosina (GC). Estas enzimas requerem a hidrólise do ATP. À medida que o ADN se abre, surgem estruturas em forma de Y chamadas bifurcações de replicação São formados dois garfos de replicação na origem da replicação, permitindo a replicação bidirecional e a formação de uma estrutura que se assemelha a uma bolha quando observada com um microscópio eletrónico de transmissão; como resultado, esta estrutura é designada por bolha de replicação O ADN perto de cada forquilha de replicação é revestido com proteínas de ligação de cadeia simples para evitar que o ADN de cadeia simples se enrole numa dupla hélice.
Uma vez que o ADN de cadeia simples está acessível na origem da replicação, a replicação do ADN pode começar. No entanto, a DNA pol III só é capaz de adicionar nucleótidos na direção 5′ a 3′ (uma nova cadeia de ADN só pode ser estendida nesta direção). Isto deve-se ao facto de a DNA polimerase necessitar de um grupo 3′-OH livre ao qual pode adicionar nucleótidos através da formação de uma ligação fosfodiéster covalente entre a extremidade 3′-OH e a extremidade 5′Isto também significa que não pode adicionar nucleótidos se não estiver disponível um grupo 3′-OH livre, o que é o caso de uma única cadeia de ADN. O problema é resolvido com a ajuda de uma sequência de ARN que fornece a extremidade 3′-OH livre. Uma vez que esta sequência permite o início da síntese de ADN, é apropriadamente designada por cartilha O iniciador tem cinco a 10 nucleótidos de comprimento e é complementar ao ADN parental ou molde. É sintetizado pelo ARN primase , que é um RNA polimerase Ao contrário das DNA polimerases, as RNA polimerases não precisam de um grupo 3′-OH livre para sintetizar uma molécula de RNA. Agora que o primer fornece o grupo 3′-OH livre, a DNA polimerase III pode agora estender este primer de RNA, adicionando nucleotídeos de DNA um a um que são complementares à fita molde (Figura 1).
Alongamento
Durante alongamento na replicação do ADN A adição de nucleótidos ocorre à sua taxa máxima de cerca de 1000 nucleótidos por segundo. A ADN polimerase III só pode estender-se na direção 5′ a 3′, o que coloca um problema na forquilha de replicação. A dupla hélice do ADN é antiparalela; isto é, uma cadeia está orientada na direção 5′ a 3′ e a outra está orientada na direção 3′ a 5′ (ver Estrutura e Função do ADN). Durantereplicação, uma fita, que é complementar à fita de DNA parental de 3 'a 5', é sintetizada continuamente em direção à forquilha de replicação porque a polimerase pode adicionar nucleotídeos nessa direção. Esta fita continuamente sintetizada é conhecida como fio condutor A outra cadeia, complementar ao ADN parental de 5′ a 3′, afasta-se da forquilha de replicação, pelo que a polimerase tem de voltar a aproximar-se da forquilha de replicação para começar a adicionar bases a um novo iniciador, novamente na direção afastada da forquilha de replicação. Fá-lo até esbarrar na cadeia previamente sintetizada e depois volta a afastar-se (Figura 4). Estes passos produzem pequenos fragmentos de ADNfragmentos de sequência conhecidos como Fragmentos de Okazaki Os fragmentos Okazaki têm o nome da equipa de investigação japonesa e do casal Reiji e Tsuneko Okazaki A vertente com os fragmentos de Okazaki é conhecida como fio de atraso e diz-se que a sua síntese é descontínua.
A cadeia principal pode ser estendida a partir de um único iniciador, ao passo que a cadeia retardatária necessita de um novo iniciador para cada um dos fragmentos curtos de Okazaki. A direção geral da cadeia retardatária será de 3′ a 5′, e a da cadeia principal de 5′ a 3′. braçadeira deslizante A pinça deslizante é uma proteína em forma de anel que se liga ao ADN e mantém a polimerase no lugar. Para além do seu papel na iniciação, topoisomerase também evita o enrolamento da dupla hélice de ADN antes da forquilha de replicação, à medida que o ADN se vai abrindo; fá-lo provocando cortes temporários na hélice de ADN e voltando a selá-la. À medida que a síntese prossegue, os primers de ARN são substituídos por ADN. Os primers são removidos pela exonuclease As fendas que permanecem entre o ADN recém-sintetizado (que substituiu o iniciador de ARN) e o ADN previamente sintetizado são seladas pela enzima DNA ligase que catalisa a formação de uma ligação fosfodiéster covalente entre a extremidade 3′-OH de um fragmento de ADN e a extremidade 5′ fosfato do outro fragmento, estabilizando a espinha dorsal açúcar-fosfato da molécula de ADN.
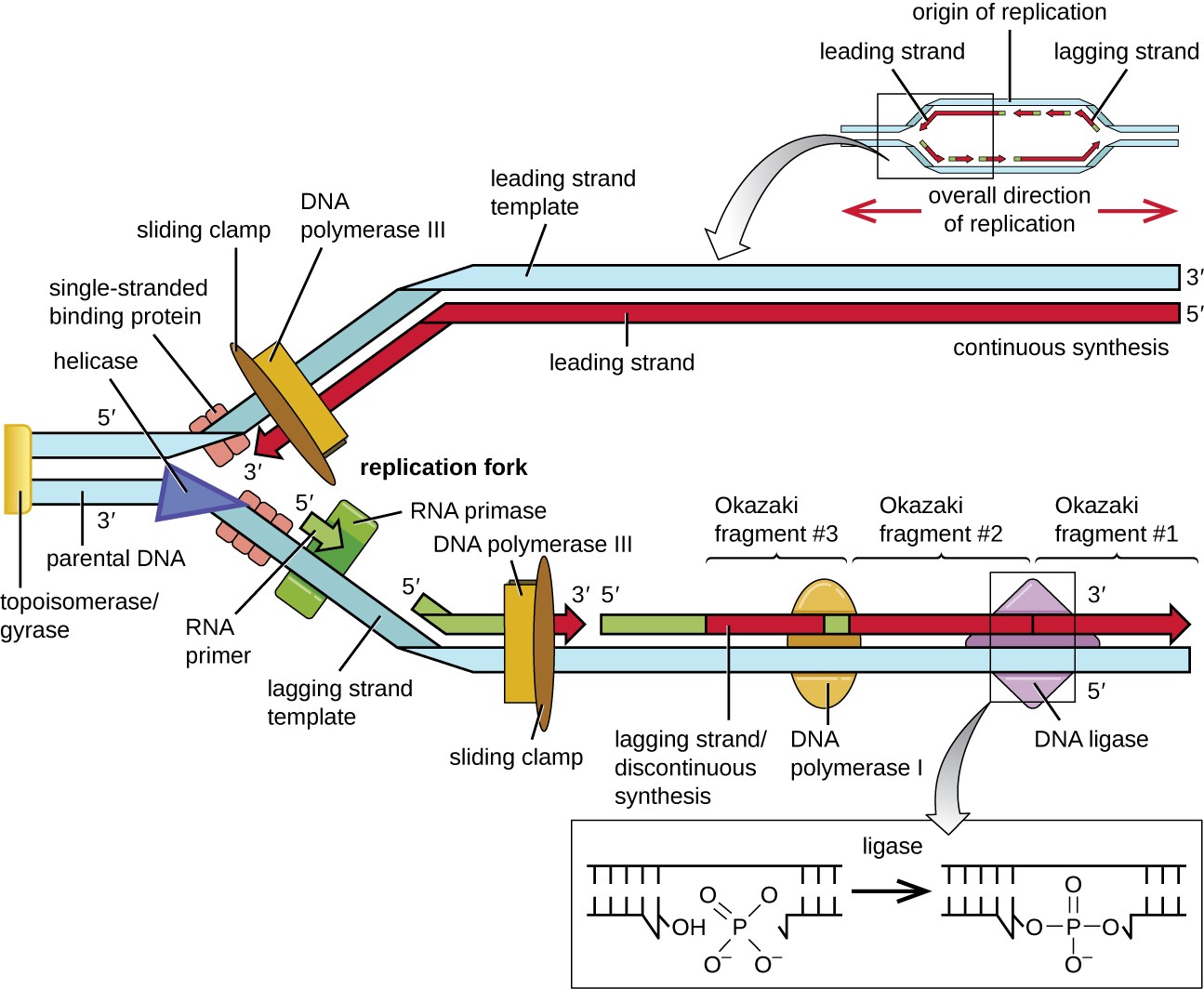
Figura 4. Clique para ampliar a imagem. Na origem da replicação, a topoisomerase II relaxa o cromossoma enrolado. Duas forquilhas de replicação são formadas pela abertura do ADN de cadeia dupla na origem, e a helicase separa as cadeias de ADN, que são revestidas por proteínas de ligação de cadeia simples para manter as cadeias separadas. A replicação do ADN ocorre em ambas as direcções. Um iniciador de ARNcomplementar à cadeia parental é sintetizada pela RNA primase e é alongada pela DNA polimerase III através da adição de nucleótidos à extremidade 3′-OH. Na cadeia principal, o DNA é sintetizado continuamente, enquanto que na cadeia posterior, o DNA é sintetizado em pequenos trechos denominados fragmentos de Okazaki. Os primers de RNA na cadeia posterior são removidos pela atividade de exonuclease do DNApolimerase I, e os fragmentos de Okazaki são unidos pela DNA ligase.
Rescisão
Uma vez replicado o cromossoma completo, terminação da replicação do ADN Embora se saiba muito sobre o início da replicação, sabe-se menos sobre o processo de terminação. Após a replicação, os genomas circulares completos resultantes dos procariotas são concatenados, o que significa que os cromossomas circulares de ADN estão interligados e devem ser separados uns dos outros. Isto é conseguido através da atividade da topoisomerase IV bacteriana, que introduzA enzima de replicação de ADN de cadeia dupla quebra as moléculas de ADN, permitindo que estas se separem umas das outras; a enzima volta então a selar os cromossomas circulares. A resolução de concatéres é um problema exclusivo da replicação do ADN procariótico devido aos seus cromossomas circulares. DNA girase e topoisomerase IV são distintas das suas congéneres eucarióticas, estas enzimas servem de alvo a uma classe de medicamentos antimicrobianos denominados quinolonas .
Reserve algum tempo para analisar o Quadro 1.Pensar no assunto
- Que enzima quebra as ligações de hidrogénio que mantêm unidas as duas cadeias de ADN para que a replicação possa ocorrer?
- É a cadeia de atraso ou a cadeia de avanço que é sintetizada na direção da abertura da forquilha de replicação?
- Que enzima é responsável pela remoção dos primers de ARN no ADN bacteriano recentemente replicado?
Replicação do ADN em eucariotas
Os genomas eucarióticos são muito mais complexos e maiores do que os genomas procarióticos e são tipicamente compostos por múltiplos cromossomas lineares (Quadro 2). genoma humano Existem múltiplas origens de replicação em cada cromossoma eucariótico (Figura 5); o genoma humano tem 30 000 a 50 000 origens de replicação. A taxa de replicação é de aproximadamente 100 nucleótidos por segundo - 10 vezes mais lenta do que a replicação procariótica.
| Tabela 2: Comparação da replicação bacteriana e eucariótica | ||
|---|---|---|
| Imóveis | Bactérias | Eucariotas |
| Estrutura do genoma | Cromossoma circular único | Cromossomas lineares múltiplos |
| Número de origens por cromossoma | Individual | Múltiplos |
| Taxa de replicação | 1000 nucleótidos por segundo | 100 nucleótidos por segundo |
| Telomerase | Não presente | Presente |
| Remoção do iniciador de ARN | DNA pol I | RNase H |
| Alongamento da cadeia | DNA pol III | pol δ, pol ε |
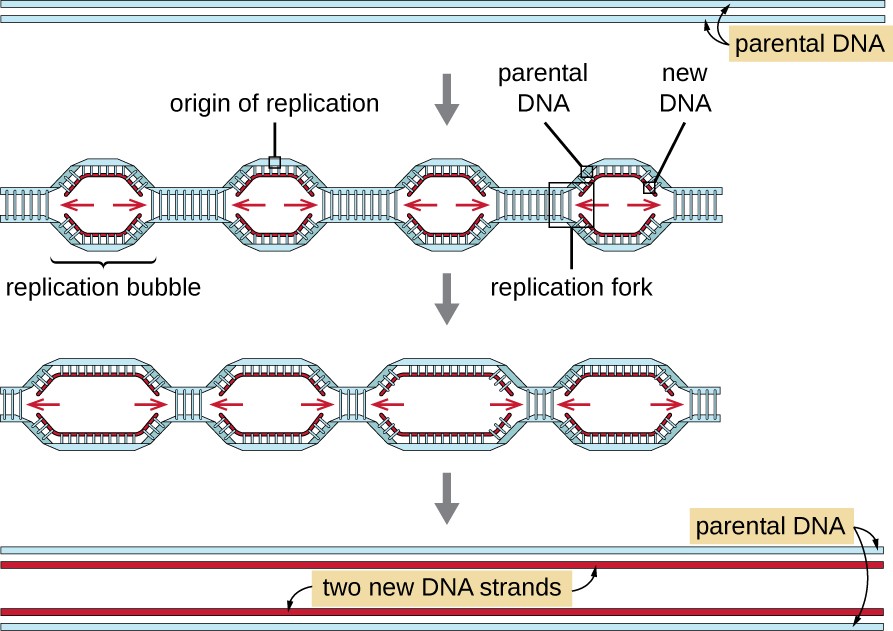
Figura 5. Os cromossomas eucarióticos são tipicamente lineares, e cada um contém múltiplas origens de replicação.
Os passos essenciais da replicação nos eucariotas são os mesmos que nos procariotas. Antes de a replicação poder começar, o ADN tem de ser disponibilizado como modelo. O ADN eucariótico é altamente enrolado e empacotado, o que é facilitado por muitas proteínas, incluindo histonas (ver Estrutura e função dos genomas celulares). origem da replicação , um complexo de pré-replicação composto por várias proteínas, incluindo helicase A topoisomerase forma e recruta outras enzimas envolvidas no início da replicação, incluindo a topoisomerase para relaxar o superenrolamento, a proteína de ligação de cadeia simples, RNA primase e ADN polimerase Após o início da replicação, num processo semelhante ao encontrado nos procariotas, o alongamento é facilitado pelas polimerases de ADN eucarióticas. A cadeia principal é continuamente sintetizada pela enzima pol δ da polimerase eucariótica, enquanto a cadeia mais atrasada é sintetizada pela pol ε. Uma proteína de fixação deslizante mantém a polimerase de ADN no lugar para que não caia do ADN. A enzimaribonuclease H ( RNase H ), em vez de uma polimerase de ADN como nas bactérias, remove o iniciador de ARN, que é então substituído por nucleótidos de ADN. As lacunas que permanecem são seladas por DNA ligase .
Uma vez que os cromossomas eucarióticos são lineares, seria de esperar que a sua replicação fosse mais simples. Tal como nos procariotas, a polimerase do ADN eucariótico só pode adicionar nucleótidos na direção 5′ a 3′. Na cadeia principal, a síntese continua até atingir o fim do cromossoma ou outra forquilha de replicação que progrida na direção oposta. Na cadeia secundária,Quando a forquilha de replicação chega ao final do cromossoma linear, não há lugar para fazer um primer para o fragmento de ADN a ser copiado no final do cromossoma. Estas extremidades permanecem assim não emparelhadas e, ao longo do tempo, podem tornar-se progressivamente mais curtas à medida que as células continuam a dividir-se.
As extremidades dos cromossomas lineares são conhecidas como telómeros Os telómeros protegem as sequências codificantes de se perderem à medida que as células se dividem. Nos seres humanos, uma sequência de seis pares de bases, TTAGGG, é repetida 100 a 1000 vezes para formar o telómero. A descoberta da enzima telomerase (A telomerase contém uma parte catalítica e um molde de ARN incorporado. Liga-se à extremidade do cromossoma e as bases complementares ao molde de ARN são adicionadas à extremidade 3′ da cadeia de ADN. Quando a extremidade 3′ do molde da cadeia atrasada estiver suficientemente alongada, a ADN polimerase pode adicionar os nucleótidos complementaresNos seres humanos, a telomerase é tipicamente ativa nas células germinativas e nas células estaminais adultas; não é ativa nas células somáticas adultas e pode estar associada ao envelhecimento destas células. Os micróbios eucariotas, incluindo fungos e protozoários, também produzem telomerase para manter a integridade cromossómica. Pela sua descoberta daA telomerase e a sua ação, Elizabeth Blackburn (1948-) recebeu o Prémio Nobel da Medicina ou Fisiologia em 2009.
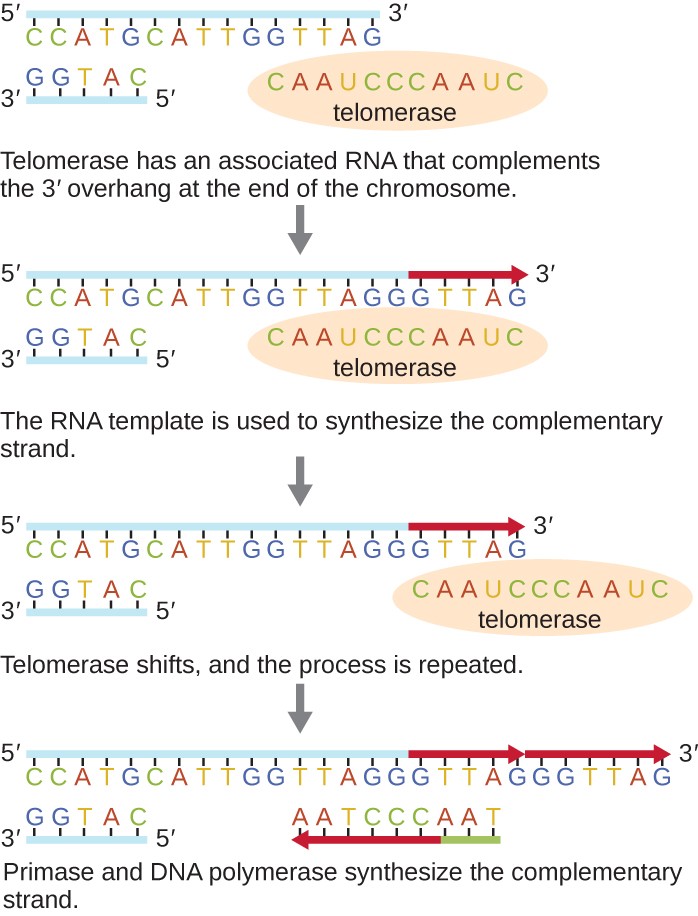
Figura 6. Clique para ampliar. Nos eucariotas, as extremidades dos cromossomas lineares são mantidas pela ação da enzima telomerase.
Esta animação ilustra o processo de replicação do ADN.Pensar no assunto
- Em que é que a origem da replicação difere entre eucariotas e procariotas?
- Que enzimas polimerase são responsáveis pela síntese de ADN durante a replicação eucariótica?
- O que se encontra nas extremidades dos cromossomas nos eucariotas e porquê?
Replicação do ADN de elementos extracromossómicos: plasmídeos e vírus
Para copiar os seus ácidos nucleicos, os plasmídeos e os vírus utilizam frequentemente variações do padrão de replicação do ADN descrito para os genomas procariotas. Para mais informações sobre a vasta gama de estratégias de replicação viral, consulte O ciclo de vida viral.
Replicação do círculo de rolamento
Enquanto muitas bactérias plasmídeos (ver Características únicas das células procarióticas) replicam-se por um processo semelhante ao utilizado para copiar o cromossoma bacteriano, outros plasmídeos, vários bacteriófagos e alguns vírus dos eucariotas utilizam replicação do círculo rolante (A natureza circular dos plasmídeos e a circularização de alguns genomas virais aquando da infeção tornam isto possível. A replicação em círculo começa com o corte enzimático de uma das cadeias da molécula circular de cadeia dupla no ponto sítio de origem de cadeia dupla (dso) Nas bactérias, a ADN polimerase III liga-se ao grupo 3′-OH da cadeia cortada e começa a replicar unidireccionalmente o ADN utilizando a cadeia não cortada como molde, deslocando a cadeia cortada à medida que o faz. A conclusão da replicação do ADN no local da quebra original resulta na deslocação total da cadeia cortada, que pode então recircular numa molécula de ADN de cadeia simples. ARNA primase sintetiza então um primer para iniciar a replicação do ADN no sítio de origem de cadeia simples (sso) da molécula de ADN de cadeia simples (ssDNA), dando origem a uma molécula de ADN de cadeia dupla (dsDNA) idêntica à outra molécula de ADN circular.
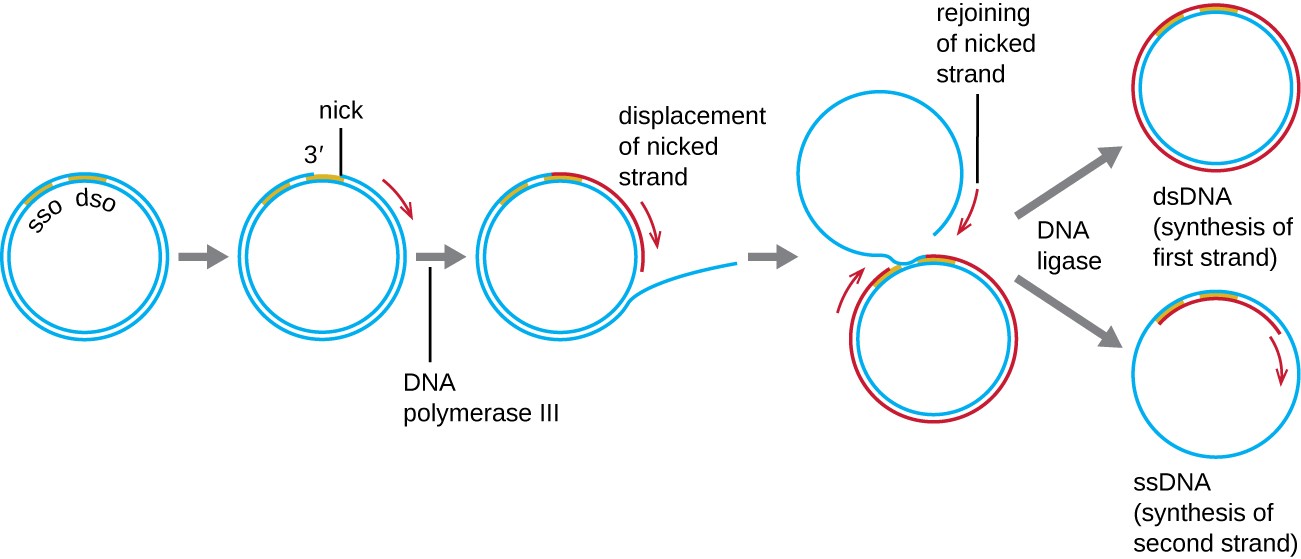
Figura 7: O processo de replicação do círculo rolante resulta na síntese de uma única cópia nova da molécula de ADN circular, como se mostra aqui.
Pensar no assunto
- Existe um fio de atraso na replicação dos círculos rolantes? Porquê ou porque não?
Conceitos-chave e resumo
- O processo de replicação do ADN é semiconservador que resulta em duas moléculas de ADN, cada uma com uma cadeia parental de ADN e uma cadeia recém-sintetizada.
- Nas bactérias, o início da replicação ocorre no origem da replicação , em que superenrolado O ADN é desenrolado por DNA girase , feita de cadeia simples por helicase e vinculado por proteína de ligação de cadeia simples para manter o seu estado de cadeia simples. Primase sintetiza um ARN curto cartilha que fornece um grupo 3′-OH livre ao qual ADN polimerase III pode adicionar nucleótidos de ADN.
- Durante alongamento , o fio condutor de ADN é sintetizada continuamente a partir de um único iniciador. fio de atraso é sintetizada de forma descontínua em curtos Fragmentos de Okazaki Os primers de ARN são removidos e substituídos por nucleótidos de ADN por bactérias ADN polimerase I e DNA ligase fecha os espaços entre estes fragmentos.
- Rescisão de replicação em bactérias envolve a resolução de concâmeros circulares de ADN pela topoisomerase IV para libertar as duas cópias do cromossoma circular.
- Os eucariotas têm tipicamente múltiplos cromossomas lineares, cada um com múltiplas origens de replicação. Em geral, a replicação nos eucariotas é semelhante à dos procariotas.
- A natureza linear dos cromossomas eucarióticos exige telómeros para proteger os genes perto da extremidade dos cromossomas. Telomerase prolonga os telómeros, impedindo a sua degradação, em alguns tipos de células.
- Replicação de círculo rolante é um tipo de síntese rápida de ADN unidirecional de uma molécula de ADN circular utilizada para a replicação de alguns plasmídeos.