Resultados da aprendizagem
- Compreender os sistemas de classificação de vírus passados e emergentes
Dado que a maioria dos vírus evoluiu provavelmente a partir de antepassados diferentes, os métodos sistemáticos que os cientistas utilizaram para classificar as células procarióticas e eucarióticas não são muito úteis. Se os vírus representam "restos" de organismos diferentes, então mesmo a análise genómica ou proteica não é útil. Porquê? Porque os vírus não têm uma sequência genómica comum que todos partilhem Por exemplo, a sequência 16S rRNA, tão útil para a construção de filogenias de procariotas, não tem qualquer utilidade para uma criatura sem ribossomas! No passado, os biólogos utilizaram vários sistemas de classificação. Os vírus foram inicialmente agrupados pela sua morfologia comum. Mais tarde, os grupos de vírus foram classificados pelo tipo de ácido nucleico que continham, ADN ou ARN, e se o seu ácido nucleico era simples ouNo entanto, estes métodos de classificação anteriores agrupavam os vírus de forma diferente, uma vez que se baseavam em diferentes conjuntos de caracteres do vírus. O método de classificação mais utilizado atualmente é designado por esquema de classificação de Baltimore e baseia-se na forma como o ARN mensageiro (ARNm) é gerado em cada tipo específico de vírus.
Sistemas de classificação anteriores
Os vírus contêm apenas alguns elementos pelos quais podem ser classificados: o genoma viral, o tipo de capsídeo e a estrutura do envelope para os vírus envelopados. Todos estes elementos foram utilizados no passado para a classificação viral (Quadro 1 e Figura 1). Os genomas virais podem variar quanto ao tipo de material genético (ADN ou ARN) e à sua organização (cadeia simples ou dupla, linear ou circular eEm alguns vírus, as proteínas adicionais necessárias para a replicação estão associadas diretamente ao genoma ou contidas no capsídeo viral.
| Quadro 1: Classificação dos vírus por estrutura e núcleo do genoma | |
|---|---|
| Classificações principais | Exemplos |
| ARN | Vírus da raiva, retrovírus |
| ADN | Vírus do herpes, vírus da varíola |
| De cadeia simples | Vírus da raiva, retrovírus |
| De cadeia dupla | Vírus do herpes, vírus da varíola |
| Linear | Vírus da raiva, retrovírus, herpesvírus, vírus da varíola |
| Circular | Papilomavírus, muitos bacteriófagos |
| Não segmentado: o genoma é constituído por um único segmento de material genético | Vírus da parainfluenza |
| Segmentado: o genoma está dividido em vários segmentos | Vírus da gripe |
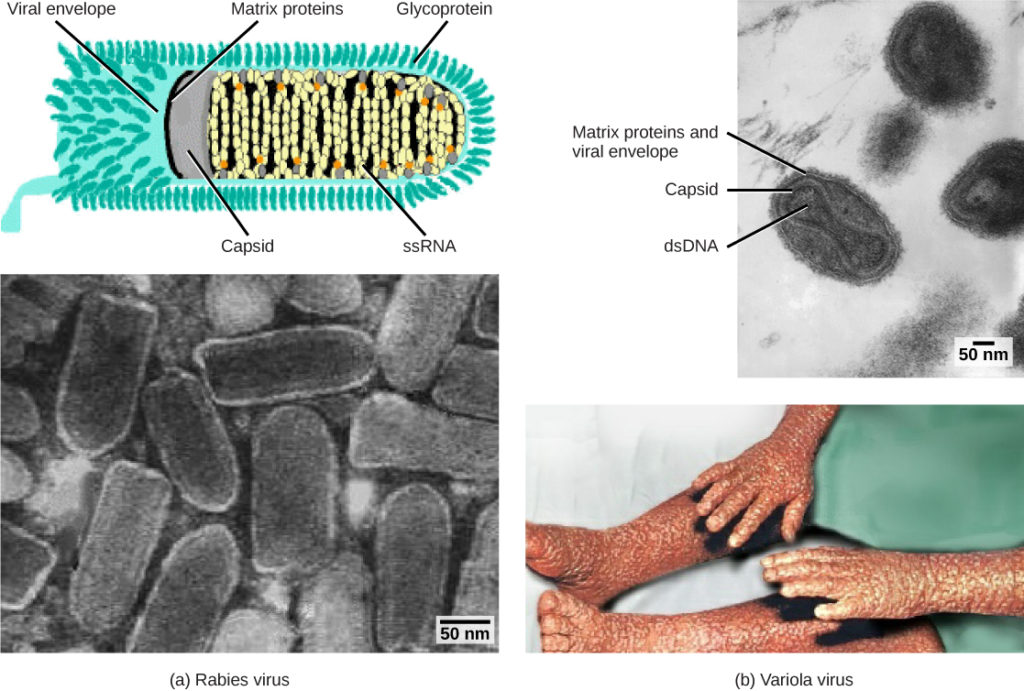
Figura 1: Os vírus são classificados com base no seu material genético central e no desenho do capsídeo. (a) O vírus da raiva tem um núcleo de RNA de cadeia simples (ssRNA) e um capsídeo helicoidal envelopado, enquanto (b) o vírus da varíola, o agente causador da varíola, tem um núcleo de DNA de cadeia dupla (dsDNA) e um capsídeo complexo. (crédito "diagrama da raiva": modificação do trabalho do CDC; "micrografia da raiva": modificação do trabalho deDr. Fred Murphy, CDC; crédito "small pox micrograph": modificação do trabalho do Dr. Fred Murphy, Sylvia Whitfield, CDC; crédito "smallpox photo": modificação do trabalho do CDC; dados da barra de escala de Matt Russell)
Os vírus também podem ser classificados de acordo com o desenho dos seus capsídeos (Quadro 2 e Figura 2). Os capsídeos são classificados como icosaédricos nus, icosaédricos envelopados, helicoidais envelopados, helicoidais nus e complexos. O tipo de material genético (ADN ou ARN) e a sua estrutura (cadeia simples ou dupla, linear ou circular, segmentada ou não segmentada) são utilizados para classificar as estruturas centrais dos vírus (Quadro 2).
| Quadro 2: Classificação dos vírus por estrutura do capsídeo | |
|---|---|
| Classificação do capsídeo | Exemplos |
| Nus icosaédricos | Vírus da hepatite A, poliovírus |
| Envolvente icosaédrica | Vírus Epstein-Barr, vírus do herpes simplex, vírus da rubéola, vírus da febre amarela, VIH-1 |
| Envolvente helicoidal | Vírus da gripe, vírus da papeira, vírus do sarampo, vírus da raiva |
| Helicoidal nua | Vírus do mosaico do tabaco |
| Complexo com muitas proteínas; alguns têm combinações de estruturas de capsídeo icosaédricas e helicoidais | Herpesvírus, vírus da varíola, vírus da hepatite B, bacteriófago T4 |
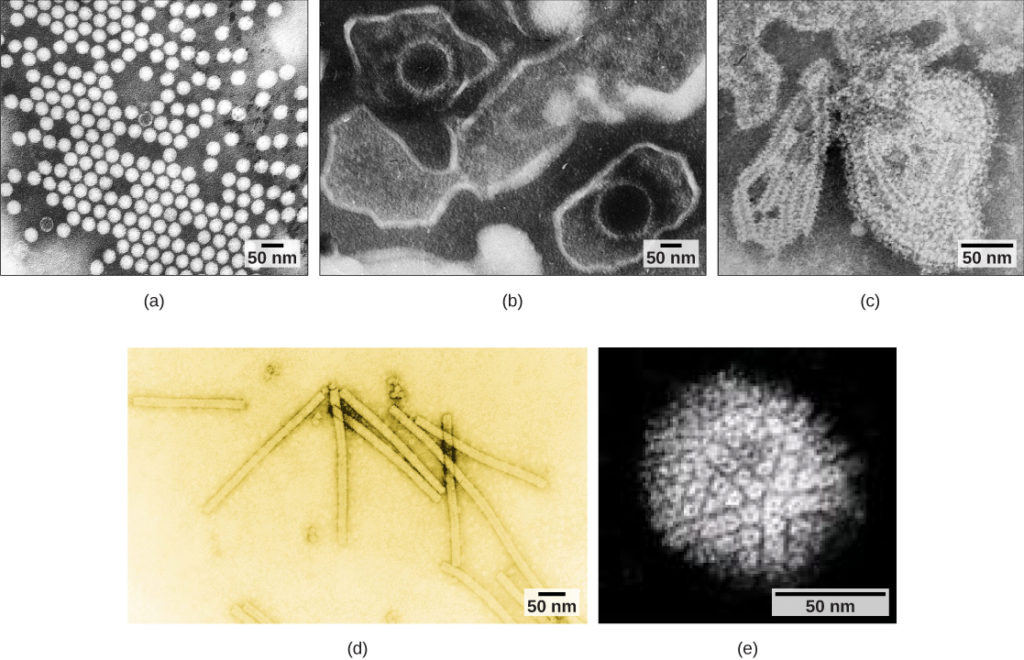
Figura 2. Micrografias electrónicas de transmissão de vários vírus mostram as suas estruturas. O capsídeo do (a) vírus da poliomielite é icosaédrico nu; (b) o capsídeo do vírus Epstein-Barr é icosaédrico envelopado; (c) o capsídeo do vírus da papeira é uma hélice envelopada; (d) o capsídeo do vírus do mosaico do tabaco é helicoidal nu; e (e) o capsídeo do herpesvírus é complexo. (crédito a: modificação do trabalho do Dr. Fred Murphy,Sylvia Whitfield; crédito b: modificação do trabalho de Liza Gross; crédito c: modificação do trabalho do Dr. F. A. Murphy, CDC; crédito d: modificação do trabalho do USDA ARS; crédito e: modificação do trabalho de Linda Stannard, Departamento de Microbiologia Médica, Universidade da Cidade do Cabo, África do Sul, NASA; dados da barra de escala de Matt Russell)
Classificação de Baltimore
O sistema de classificação de vírus mais comummente utilizado foi desenvolvido pelo biólogo David Baltimore, galardoado com o Prémio Nobel, no início da década de 1970. Para além das diferenças morfológicas e genéticas acima referidas, o esquema de classificação de Baltimore agrupa os vírus de acordo com a forma como o ARNm é produzido durante o ciclo replicativo do vírus.
Grupo I Os vírus contêm ADN de cadeia dupla (dsDNA) como genoma e o seu ARNm é produzido por transcrição da mesma forma que o ADN celular.
Grupo II Os vírus têm como genoma o ADN de cadeia simples (ssDNA) e convertem os seus genomas de cadeia simples num intermediário de dsDNA antes de poderem transcrever-se em ARNm.
Grupo III Os vírus utilizam o dsRNA como genoma. As cadeias separam-se e uma delas é utilizada como modelo para a geração de mRNA através da RNA polimerase dependente de RNA codificada pelo vírus.
Grupo IV Os vírus têm como genoma o ssRNA com uma polaridade positiva. Polaridade positiva significa que o ARN genómico pode servir diretamente como ARNm. Os intermediários do dsRNA, chamados intermediários replicativos A partir destes intermediários formam-se múltiplas cadeias completas de ARN de polaridade negativa (complementares ao ARN genómico de cadeia positiva), que podem depois servir de modelo para a produção de ARN com polaridade positiva, incluindo tanto o ARN genómico completo como os ARNm virais mais curtos.
Grupo V Os vírus contêm genomas de ssRNA com um polaridade negativa Tal como acontece com os vírus do Grupo IV, os intermediários de dsRNA são utilizados para fazer cópias do genoma e produzir mRNA. Neste caso, o genoma de cadeia negativa pode ser convertido diretamente em mRNA. Além disso, são feitas cadeias completas de RNA positivo para servir de modelo para a produção do genoma de cadeia negativa.
Grupo VI Os vírus têm genomas de ssRNA diplóides (duas cópias) que devem ser convertidos, utilizando a enzima transcriptase reversa O dsDNA é então transportado para o núcleo da célula hospedeira e inserido no genoma do hospedeiro. Em seguida, o mRNA pode ser produzido por transcrição do DNA viral que foi integrado no genoma do hospedeiro.
Grupo VII Os vírus têm genomas parciais de dsDNA e produzem intermediários de ssRNA que actuam como mRNA, mas também são convertidos novamente em genomas de dsDNA pela transcriptase reversa, necessária para a replicação do genoma. As características de cada grupo na classificação de Baltimore estão resumidas na Tabela 3, com exemplos de cada grupo.
| Tabela 3: Classificação de Baltimore | |||
|---|---|---|---|
| Grupo | Características | Modo de produção de ARNm | Exemplo |
| I | ADN de cadeia dupla | O ARNm é transcrito diretamente a partir do modelo de ADN | Herpes simplex (herpesvírus) |
| II | ADN de cadeia simples | O ADN é convertido para a forma de cadeia dupla antes de o ARN ser transcrito | Parvovírus canino (parvovírus) |
| III | ARN de cadeia dupla | O ARNm é transcrito a partir do genoma do ARN | Gastroenterite infantil (rotavírus) |
| IV | ARN de cadeia simples (+) | O genoma funciona como ARNm | Constipação comum (pircornavírus) |
| V | ARN de cadeia simples (-) | O ARNm é transcrito a partir do genoma do ARN | Raiva (rabdovírus) |
| VI | Vírus de ARN de cadeia simples com transcriptase reversa | A transcriptase reversa produz ADN a partir do genoma de ARN; o ADN é então incorporado no genoma do hospedeiro; o ARNm é transcrito a partir do ADN incorporado | Vírus da imunodeficiência humana (VIH) |
| VII | Vírus de ADN de cadeia dupla com transcriptase reversa | O genoma viral é um ADN de cadeia dupla, mas o ADN viral é replicado através de um intermediário de ARN; o ARN pode servir diretamente como ARNm ou como modelo para produzir ARNm | Vírus da hepatite B (hepadnavírus) |
Experimentar
Contribuir!
Tem alguma ideia para melhorar este conteúdo? Gostaríamos de receber a sua opinião.Melhorar esta páginaSaber mais